Conociendo Cytoscape

Importancia de Cytoscape
El avance en la última década de las ómicas ha producido un crecimiento exponencial de datos biológicos. Lo cual ha provocado la necesidad de desarrollar herramientas capaces de analizar y extraer la información más relevante de esta maraña de datos. Por muy paradójico que suene, una forma de poder sintetizar todos estos datos es a través de la generación de redes (o networks en inglés) ya que en muchas ocasiones reducen la complejidad de los datos y resultan más eficientes que las típicas tablas, son una buena estrategia para la integración de datos y permiten una visualización más intuitiva de los mismos. Además, las redes las encontramos en todas las partes (ya hemos escuchado todos hablar del famoso “social networking” 😋), especialmente en la biología como por ejemplo la comunicación celular, sistema nervioso, conexiones moleculares, etc.
Por eso hoy os vengo hablar de una herramienta muy útil en el campo de la bioinformática: Cytoscape. Se trata de un software de código abierto que sirve para visualizar redes complejas e integrarlas con cualquier otro tipo de datos. En el caso de, por ejemplo, una red genética, cada nodo representaría un gen y las aristas (la línea que une los nodos) representaría la relación existente entre ellos. Pero redes hay de muchos tipos, de genes, de proteínas, de metabolitos, metilación, rutas metabólicas, etc e incluso hay redes que combinan varios tipos de datos. Cabe añadir que Cytoscape también puede utilizarse en ámbitos no biológicos como por ejemplo para el modelado de sistemas de agua, calcular distancias entre estrellas o para el diseño de vías ferroviarias.
¿Y cómo puedo empezar a utilizar Cytoscape?
Existen muchos tutoriales tanto en Youtube como en Internet que te pueden ayudar a iniciarte en Cytoscape. Sin duda, lo que más me ayudó fue leer el propio manual de usuario de Cytoscape. Y es que ya hace algo más de un año Cytoscape y yo nos vimos las caras por primera vez, fue durante la realización de mi TFM. Y para enseñaros “a muy grosso modo” qué se puede hacer con Cytoscape, qué mejor forma que sea resolviendo, aunque muy resumidamente, el caso práctico al que me enfrenté en aquel momento:
Disponemos de datos de metabolómica de plasma (P) y de tejido cerebral (C) de ratas con una determinada enfermedad y queremos investigar si existen metabolitos en plasma que se pudieran utilizar como biomarcadores de daño cerebral, ya que actualmente la enfermedad tiene un diagnóstico tardío. Para ello se aplicó un modelo de regresión multivariante de reducción de la dimensión (PLS) donde se utilizaron los datos de plasma como matriz predictiva y los datos de cerebro como matriz respuesta.
Una vez tenemos todos los análisis hechos, el primer paso de todos es generar nuestros ficheros “inputs”. ¡Este es el paso más importante! Y es que en realidad lo más complicado no es utilizar Cytoscape en sí, sino preparar los ficheros con los que vamos a trabajar teniendo en cuenta qué queremos visualizar y cómo trabaja Cytoscape. En resumen, Cytoscape necesita dos ficheros de “input”:
- Node Table. Este fichero debe contener información relacionada con los nodos. En nuestro caso los nodos son los metabolitos, tanto de plasma como de cerebro. Por lo tanto, en la primera columna de este fichero tendrá que aparecer el nombre de todos los metabolitos (importante en este caso, ponerle una etiqueta si es de plasma [P] o de cerebro [C]). En las siguientes columnas se puede incluir atributos de cada nodo/metabolito como por ejemplo el nombre del metabolito sin la etiqueta (Metabolito), si pertenecen a plasma o al cerebro (Tejido), el coeficiente del modelo de regresión (CoefB), si están “Diferencialmente expresados” (DE), y todos los atributos que consideremos oportunos.
- Network File. En este caso, este fichero debe contener información referente a la relación existente entre los nodos (metabolitos). Que a efectos prácticos puede ser, entre otros, un coeficiente de correlación (Freq) que para que sea más sencillo de utilizar se puede codificar en función de si tiene signo negativo, positivo o es cero (signo). Las dos primeras columnas de este fichero serán los nombres de los metabolitos que estamos comparando (Var1 y Var2), importante de nuevo para este caso las etiquetas de “P_” y “C_”.
En nuestro caso los ficheros tendrían un aspecto como los siguientes:
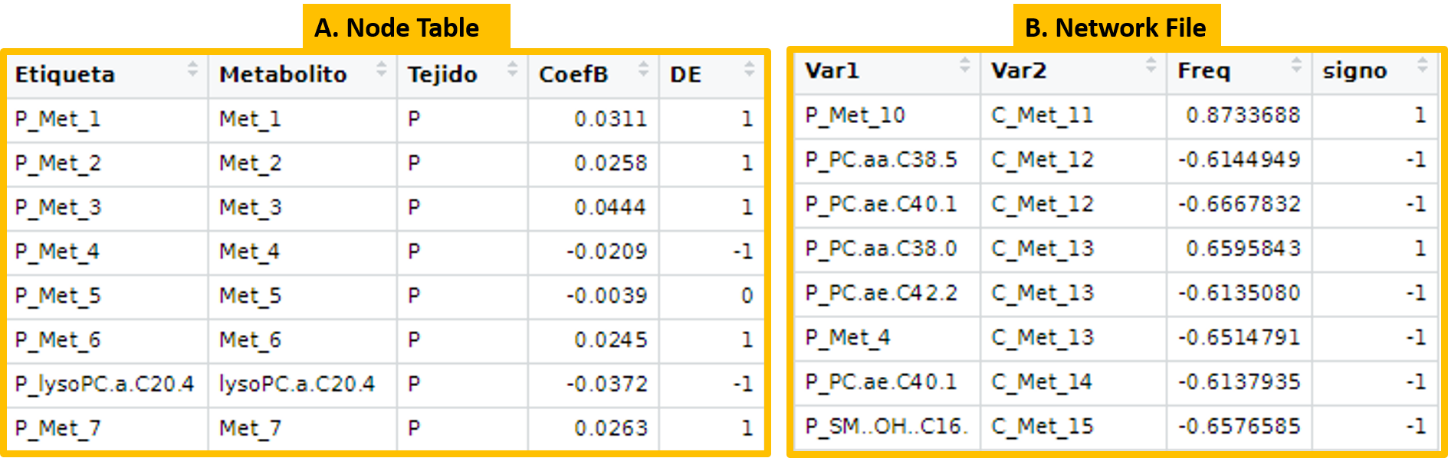
Una vez tenemos los dos ficheros de datos ya podemos empezar a utilizar Cytoscape.
Para importar nuestros propios datos tenemos que seleccionar File> Import> Network from file. Seleccionamos el fichero deseado y se nos abrirá una ventanita. Aquí tendremos que indicar cuál es nuestro 🟢 Nodo fuente (Source node) y el 🎯Nodo objetivo (Target node).
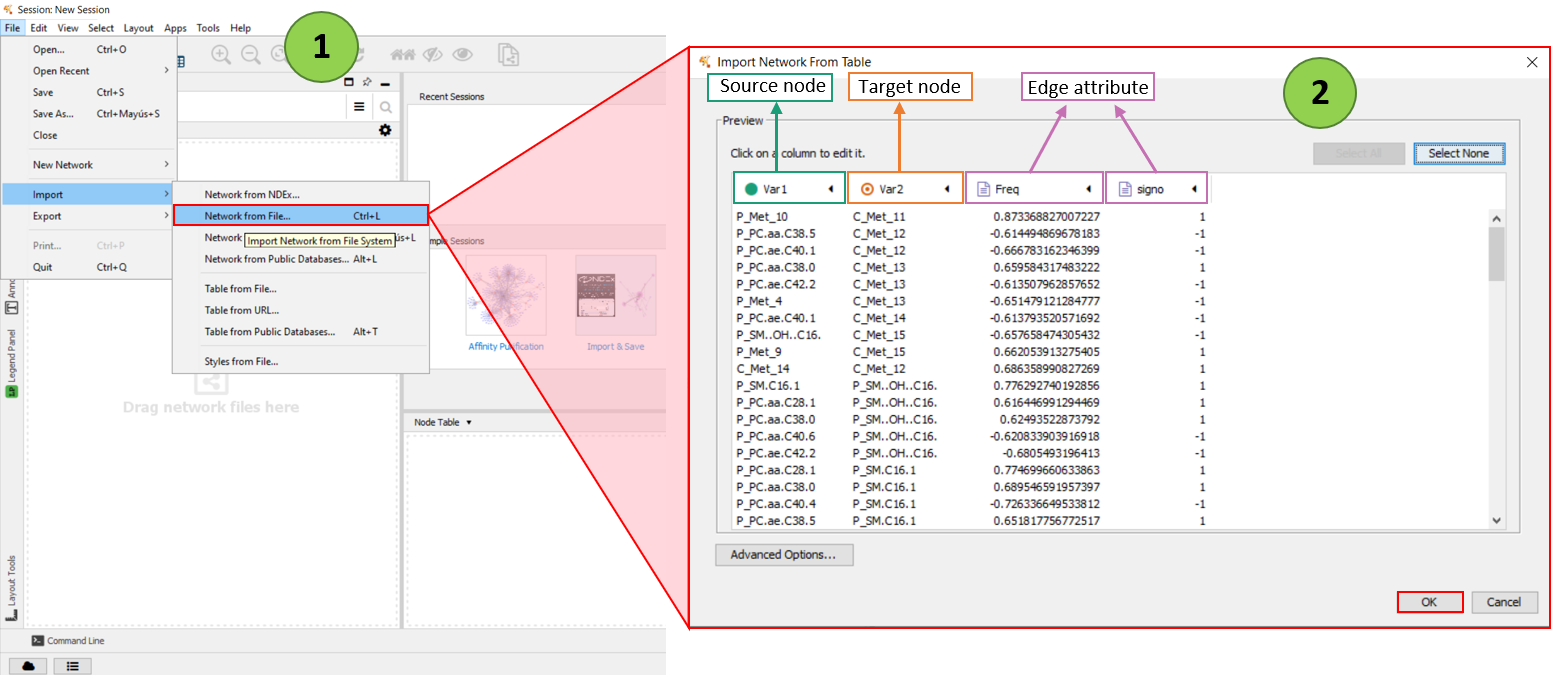
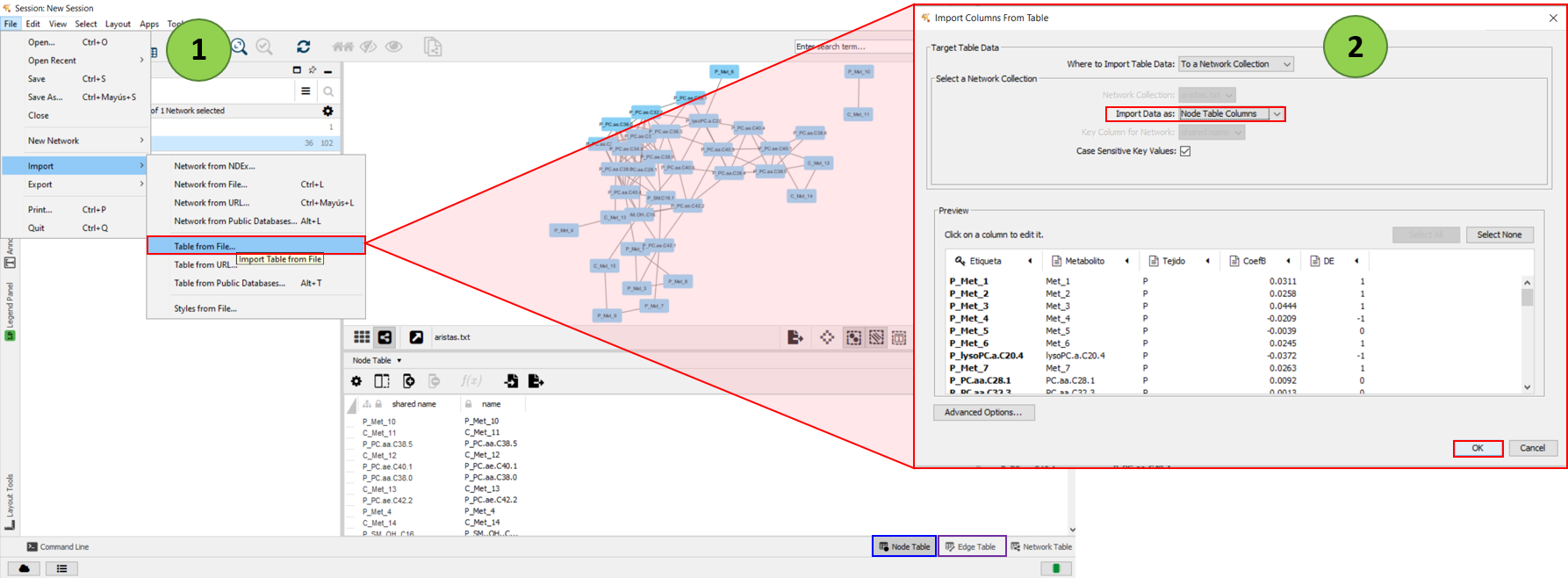
Al aceptar veremos que se genera la red. En la pestaña de “Edge Table” aparecen todos los atributos del fichero que hemos importado. Sin embargo, en la pestaña adyacente, “Node table”, no aparecen atributos. Por lo que tenemos que importar el fichero de los nodos con sus correspondientes atributos de la misma manera que hemos hecho anteriormente.
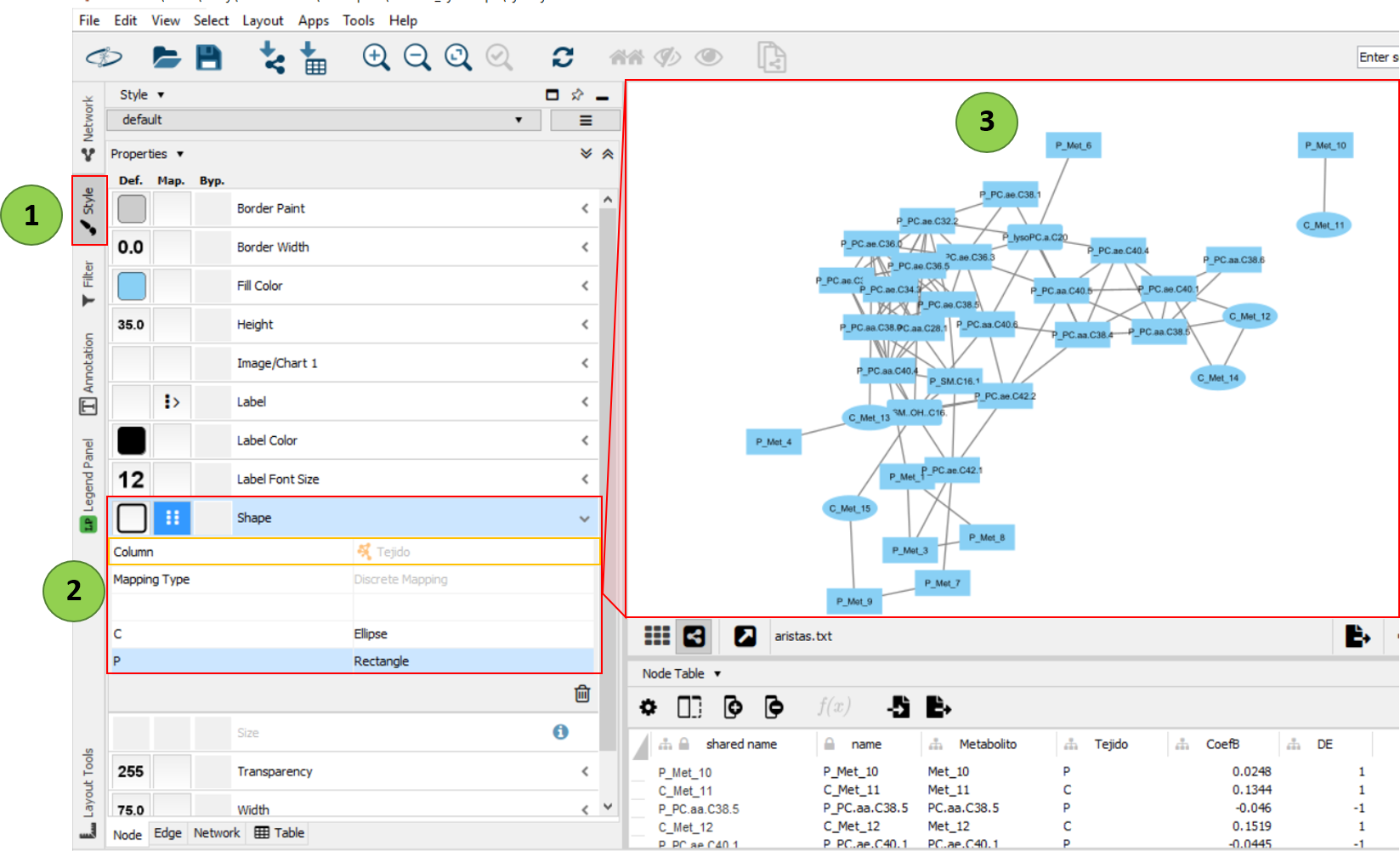
Para poder distinguir a simple vista qué metabolitos son del cerebro y cuáles del plasma, vamos a “Style” del menú de la izquierda y en la opción de “Shape”” indicamos que queremos que la forma de los nodos sea en función de la columna “Tejido”. Entonces ya nos saldrán todas las opciones existentes para esa columna, en nuestro caso C= Cerebro y P = Plasma. A continuación, ya podemos seleccionar cómo queremos que se represente cada tejido. En este ejemplo los metabolitos en plasma (P) y los de cerebro (C) los hemos representado como un rectángulo y como una elipse respectivamente.
Como ya tenemos asociada cada forma al tejido, entonces ya podríamos dejar de utilizar los prefijos/etiquetas de “P_” y “C_” y que aparezca únicamente el nombre del metabolito. Para eso vamos a “Style” del menú de la izquierda y en la opción de “Label”” indicamos que queremos que la forma de nuestra red sea en función de la columna “Metabolito”.

También podemos indicar si los metabolitos están “down o up regulated”:
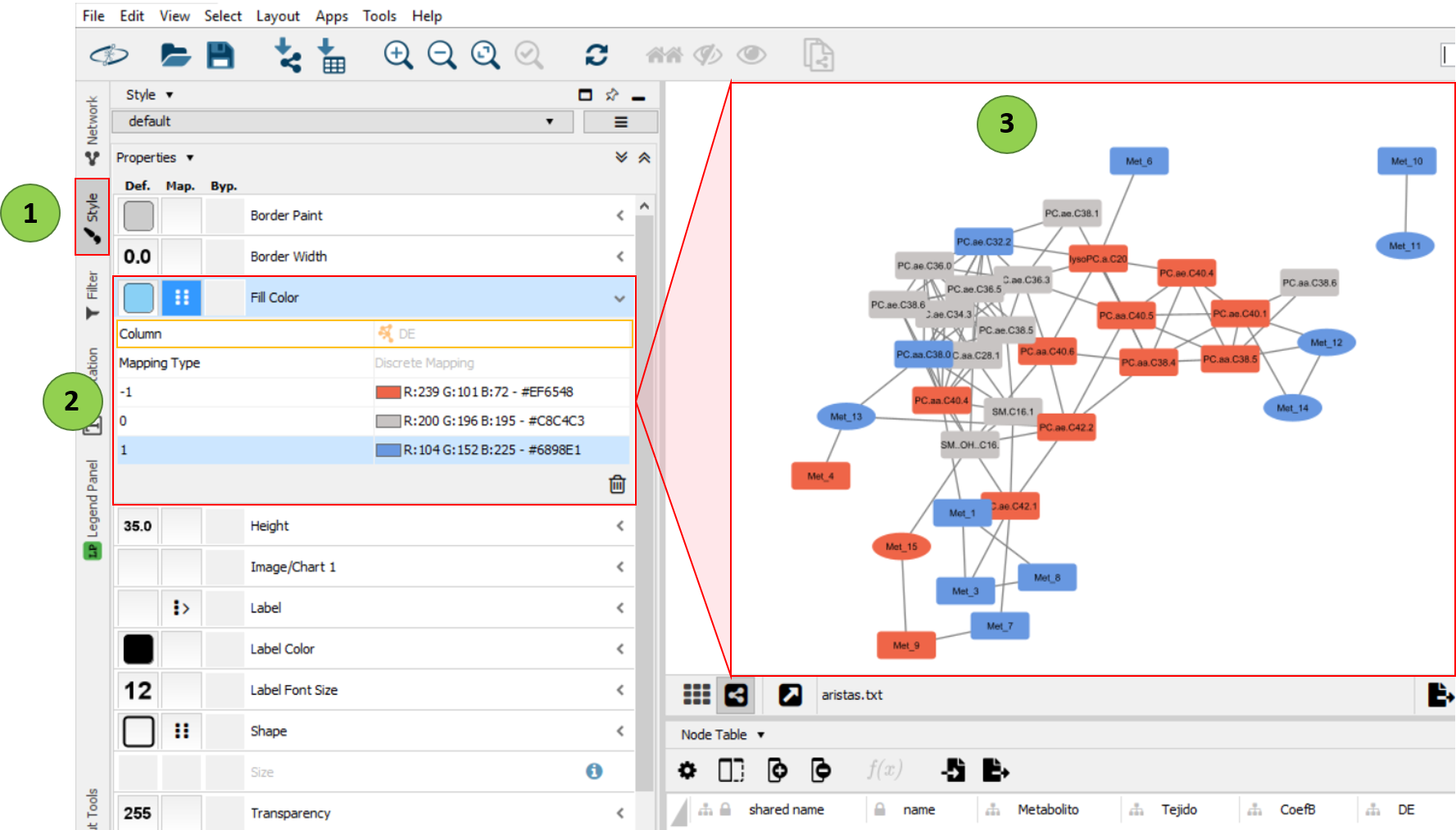
De la misma forma, podemos colorear las aristas en función de si los metabolitos están positiva o negativamente correlacionados.
Al final la red tiene que quedar lo más legible posible para el lector por ello hay que ordenarla. En la pestaña de “Layout” del menú superior, existen muchas opciones que permite realizar diferentes distribuciones de la red. Al mismo tiempo, también puedes mover los nodos individualmente si seleccionas con el ratón encima de uno de ellos y posteriormente lo mueves hasta donde desees.
Siguiendo todas estas indicaciones al final quedaría una red como la que se muestra a continuación en la que ya solo quedaría exportar la imagen.
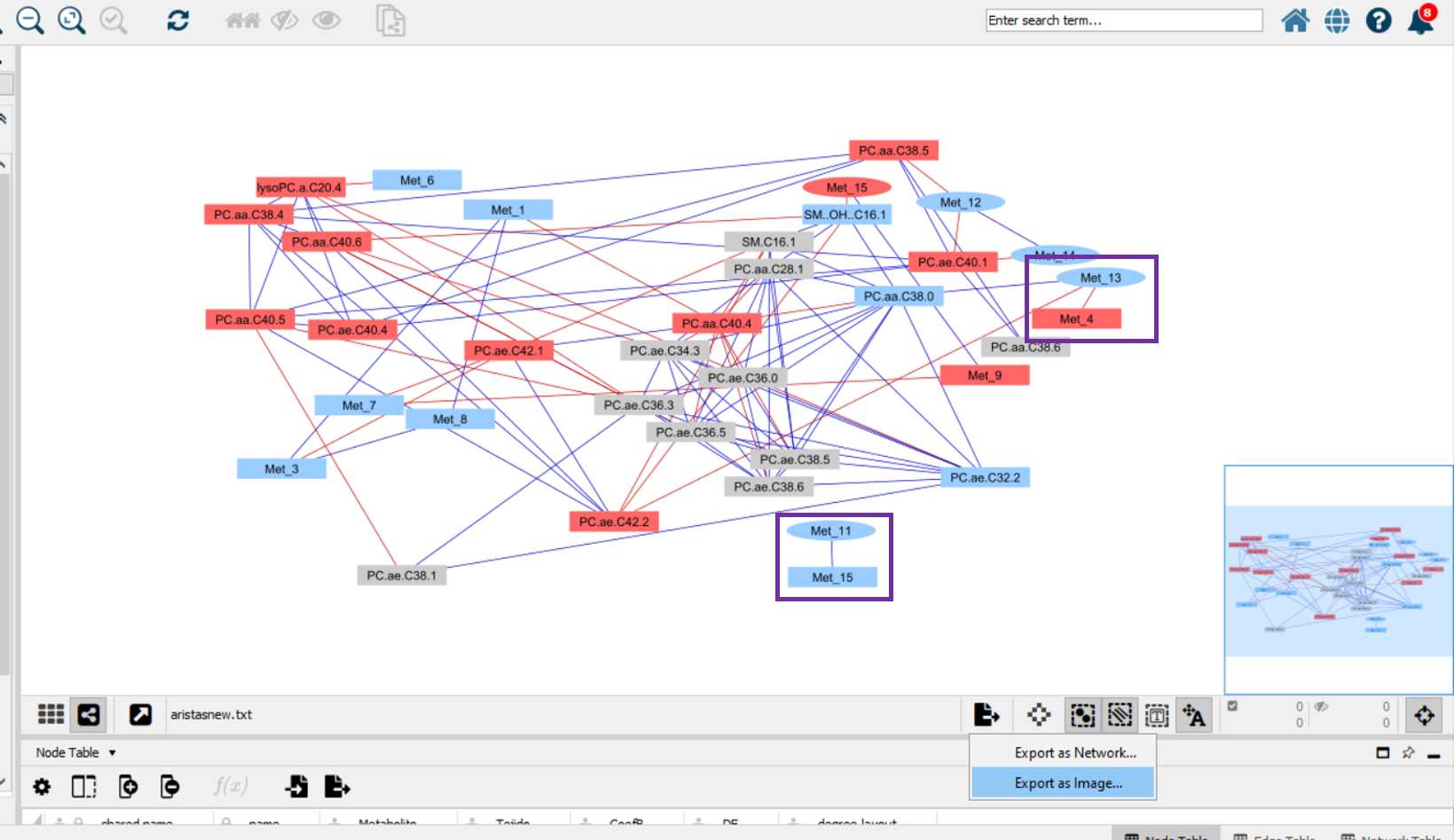
En la imagen se puede apreciar a simple vista biomarcadores candidatos en plasma de daño cerebral (cuadrados morados). Por ejemplo, se puede observar que existe una correlación positiva entre los niveles en plasma de Met_15 y los niveles en cerebro de Met_11.
Esto sería una forma de implementar Cytoscape con tus propios datos (aunque sean poquitos como en este caso). Como vemos, así hemos conseguido integrar los datos (tanto los metabolitos del plasma como del cerebro) y que nos sea más fácil e intuitivo visualizar posibles biomarcadores en plasma de daño cerebral de la enfermedad que estamos estudiando. Para ello, me repito, lo más importante aquí es saber qué quieres hacer con el programa y en función de eso elaborar tus ficheros de input.
Cytoscape ++
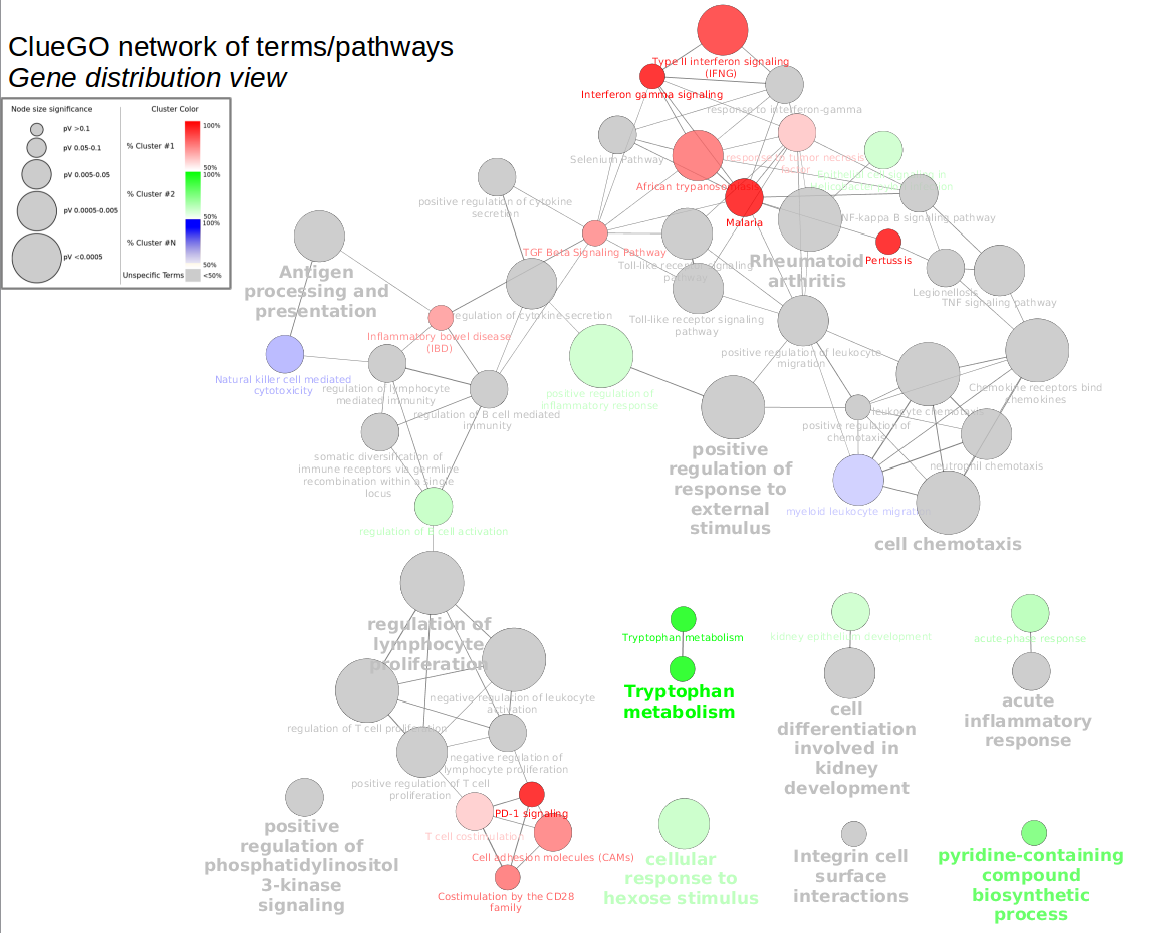
Lo que os acabo de mostrar es un ejemplo muy sencillo del uso de Cytoscape ya que este software tiene mil posibilidades gracias a los plugins y a las apps, extensiones de Cytoscape, que incorporan nuevas funciones. Los plugins y apps los puedes descargar desde Cytoscape y generalmente suelen ser gratuitos para fines académicos. Por ejemplo, uno de los plugins que más estoy utilizando últimamente son ClueGo y Clupedia, con el que se pueden realizar, entre otras cosas, análisis de enriquecimiento funcional y visualización de redes como la que se observa a continuación:
Otro ejemplo de la importancia de Cytoscape en el ámbito científico es el hecho de que Bioconductor ha incorporado un paquete R, “RCy3” que permite comunicarse con Cytoscape. La verdad que es un paquete muy útil ya que permite automatizar los procesos y te facilita la vida especialmente si ya estás trabajando desde R. Además, Cytoscape permite importar datos de repositorios públicos como IntAct, Reactome, WikiPathways, PSICQUIC, etc, con las que puedes trabajar directamente y/o implementarlas en tus estudios.
Por último, comentaros que Cytoscape es una herramienta que está teniendo una considerable implicación hoy en día en investigaciones científicas en múltiples campos como en el estudio del perfil molecular y las vías implicadas de dos tipos de cáncer cerebral, interacciones genéticas del genoma de la levadura e incluso para estudiar las proteínas humanas con las que interactúa el SARS-COV-2 para la reutilización de fármacos ya aprobados.
Como veis, Cytoscape es una herramienta muy versátil de la que no dejas día a día de aprender, solo hay que ir explorando la gran variedad de posibilidades y servicios que ofrece. En lo personal, tener ciertos conocimientos de Cytoscape me ha abierto bastantes puertas a la hora de establecer colaboraciones en estudios y entender, aunque sea de forma modesta, el increíble mundo de las redes de regulación. Es por eso que espero que este post os haya resultado útil y sobre todo os haya animado a seguir conociendo el mundo de las networks y en concreto de Cytoscape. Si has llegado hasta aquí, seguro que tenéis muchas cosas interesantes que contarme. Por eso os comparto mi contacto para que me déis un poco de feedback y compartáis conmigo todas vuestras dudas e inquietudes con respecto a este post.
